 医学资讯
医学资讯
 2021-02-04
2021-02-04
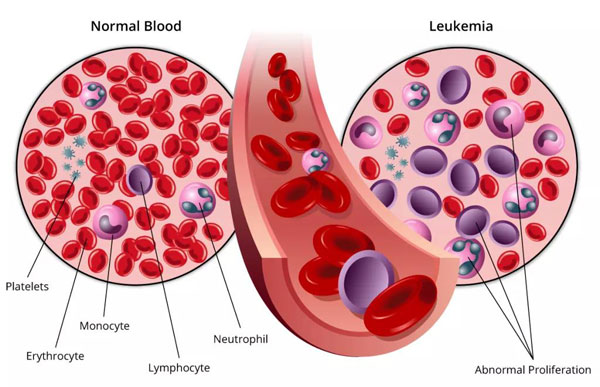
急性髓系白血病(AML)是最常见的白血病类型之一,多发生于65岁以上中老年人,疾病进展迅速,是最具侵袭性和最难治疗的血癌之一,堪称中老年人的“噩梦”。
尽管现有疗法已有了很大进步,但确诊AML的患者5年生存率仍不足30%。
目前,AML主要以强力诱导化疗为主,但AML很容易耐药,病情会迅速恶化,而且因年龄或并发症等因素,很多患者不符合造血干细胞移植条件,可选治疗方案十分有限。
▌破解急性髓系白血病耐药难题
近日,在62届美国血液学会虚拟年会上,好医友战略合作伙伴、美国肿瘤精准医学领域领军企业Cellworks公布了一项重磅临床研究(myCare-021-02)结果。
研究发现:使用TRI进行基因组学生物模拟,可通过结合患者生物标志物来改善预后,并为已发生耐药的AML患者推荐新的治疗方案。
TRI依靠来自肿瘤细胞的多组学输入,来预测和识别针对每个患者肿瘤的独特分子机制的潜在治疗方案。
AML患者特异性耐药机制的发现,为基于深层分子诊断的治疗提供了新的突破,可辅助临床治疗决策,降低无效治疗风险,提高用药疗效,降低治疗成本。
“AML是目前白血病死亡的主要原因,但使用TRI个性化治疗方案可改善患者预后。”
好医友医疗网肿瘤精准医学专家、myCare-021-02临床试验主要研究者迈克尔·卡斯特罗博士(曾多次获评“美国顶级医生”)说:
“诱导化疗反应和疾病缓解情况因肿瘤的生物亚型和用于诱导治疗的药物而异,但即使在特定的生物亚组中,反应也是不可预测的。TRI生物模拟平台提供了一种针对患者特定疾病生物标志物和耐药机制的个性化医学方法。”
“AML诱导化疗失败并不罕见,即使是在那些对治疗敏感的生物亚型中也是如此。”
田纳西大学健康科学中心教授斯科特·霍华德博士说,“TRI可识别标准诱导方案失败的原因,并根据耐药患者中共有的基因组异常给出替代疗法。”
▌关于myCare-021-02临床研究
该研究旨在预测AML患者对诱导化疗方案的反应,并确定难治性患者的基因组特征、耐药机制及预测个性化治疗选择。
这项研究从PubMed出版物中选择了57名已知对治疗有反应的AML患者。这些患者被分为3组:CBFB-MYH11融合组、RUNX1-RUNX1T1融合组和CEBPα融合组。
NCCN为有这些突变的急性髓系白血病患者推荐了具体治疗建议,对这些突变进行诱导治疗后,实现了高缓解率。
研究者使用TRI生物模拟平台分析每个图谱的可用基因组数据,以使用PubMed和其他在线资源的公开信息生成AML亚型特异性蛋白质网络图,根据患者的特异性生物标志物分析药物敏感性和耐药性通路。
针对三个队列中的每个患者肿瘤分子特征,模拟从数字药物库中筛选的药物的反应情况。通过测试每种药物对细胞生长评分(综合增殖、生存能力和凋亡指数得出)的影响来评估临床获益。
研究结果发现,在57名高危AML患者中,有6名(11%)出现诱导治疗失败;1/19携带CBFB-MYH11融合蛋白的患者对诱导治疗没有反应。
TRI确定了发生耐药的患者中导致耐药的共同基因异常:这些患者携带6号染色体三体,并有GSTA1、GSTA2、GSTA4、DEK、TFAP2A、NFYA和EHMT2基因扩增,相关信号通路异常导致了治疗失败。
TRI还确定了3种针对该患者特异性生物标志物的前瞻性治疗方案。
同样,2/10有RUNX1-RUNX1T1基因融合的患者对诱导治疗没有反应。TRI发现,EZH2中的功能缺失突变导致HOXA5和HOXA9水平升高,是治疗失败的关键因素。
此外,TRI还为该患者确定了3种针对其特异性耐药机制的前瞻性治疗方案。
在CEBPα队列中,3/28的患者对诱导治疗没有反应。对于其中一名患者,TRI发现,DNMT3A功能缺失突变和HOXA5和HOXA9水平升高,是治疗失败的关键因素。
同样,TRI也为该患者确定了3种针对其特异性生物标志物的新治疗方案。
▌TRI“沙盘推演”,让肿瘤治疗“心中有数
肿瘤治疗应答指数(TRI)依托计算机生物模拟和机器学习人工智能技术,突破性运用患者全外显子信息建立个性化疾病模型,进行生物模拟,分析基因组、蛋白质组、转录组和表观基因组变异信息对肿瘤细胞信号通路上下游的影响,并针对特定药物或药物组合进行计算。
这相当于在治疗前已进行了严密的“沙盘推演”,从而为患者预测最佳个性化用药方案,同时为面临治疗方案抉择的临床医生提供基于分子基因层级发病机制的决策支持。
一年多来,在全国近百家三甲医院,TRI检测已为数百名疑难肿瘤患者量身定制了匹配的精准治疗方案。
参考来源:
https://cellworks.life/news-events/press-releases/cellworks-cbm-biosimulation-identifies-genomic-causes-induction-failure